|
超並列シーケンサ(メーカー問わず)によって出された生データが公開されているデータベースなどはありますか? データの解析などを通していろいろ勉強してみたいと思っています。 |
|
ご所望のデータは、NCBI、EBI、DDBJでSequence Read Archive (SRA) としてアーカイブされています。 手前味噌ですが、SRAのデータを整理して、SRAs (survey of read archives)というサイトをつくりました。目的別、機器別、生物種別などからデータを検索できるようにしています。 http://sra.dbcls.jp/ 本家のDDBJでもデータの検索ができますし、何より日本語でドキュメントがあったりするのでそちらも参照されるとよいと思います。 http://trace.ddbj.nig.ac.jp/dra/index.shtml |
|
CLC Bioが"Next Generation Sequencing example data"として,次世代シーケンサで読まれたデータを幾つか公開しています.本来はCLC Genomics Workbench用のテストデータですが,Raw dataなど幾つか使える物がありそうです. http://www.clcbio.com/index.php?id=1290 Raw dataではRoche/454,Illumina/Solexa,AB/SOLiDと主要なシーケンサからのデータが揃っているほか,RNA-Seqのデータ(CLC Genomics Workbenchで読み込む形式のみ)も公開されています. 公開ライセンスはちょっとわかりませんが,個人的にNGS解析の練習などに使う分には問題ないかと思います. 自分で触ったことがないので期待にそえるデータかどうか分かりませんが,こんなデータもあるということで質問に回答してみました. |
|
私はよくNCBIのSequence Read Archive(以下SRA)を使います。 NCBIのトップページの上のボックスから"SRA"を選択して、RNA-SeqとかChIP-Seqとかの キーワードを入力するとたくさんヒットすると思います。 また多くの研究者は興味を持った論文からそのデータを取得することが多いとは思いますが、 通常論文にはSRAのIDは書かれていないことが多いです(親切な著者は書いていますが・・・)。 たぶんGene Expression Omnibus(GEO)のIDが書かれていることが多いでしょう。 この場合は同様にトップページのボックスから"GEO DataSet"を選び、GEO IDで検索をすると ヒットします。GEOに登録されている次世代シーケンサデータはマッピング後のデータ(BEDファイルなど) ですが、下の方にSRAのリンクが張ってあるので、それをたどって生データを取得できます。 また生データも年々容量が増加傾向にあり、ダウンロードに時間がかかります。 (Asperaというソフトをインストールする必要があります) |
|
SRAのデータアクセスをRでおこなうBioconductorパッケージ SRAdbというものがあります。 これは、SRAのメタデータを格納したSQLiteのファイルをNCIの方が公開していて、それを操作する形になっています。詳しくはマニュアルのPDF書類をご覧ください。 簡単な操作とコードを例示します: メタデータのダウンロードと準備、テーブル操作library(SRAdb) sqlfile <- getSRAdbFile() #メタデータをダウンロード sra_con <- dbConnect(SQLite(), sqlfile) sra_tables <- dbListTables(sra_con) # テーブル名のリストを取得 dbListFields(sra_con, "study") # "study" テーブルのフィールド名の取得 sqliteQuickSQL(sra_con, "PRAGMA TABLE_INFO(study)") # "study" テーブルの情報を一覧 SQLの実行:"study" テーブルから三つのレコードを表示rs <- dbGetQuery(sra_con, "select * from study limit 3") 変換:experimentやrunアクセッションの変換conversion <- sraConvert(c("SRP001007", "SRP000931"), sra_con = sra_con)
全文検索:"run" と "study" テーブルについて文字列 "breast" と "cancer" の含まれるエントリの検索rs <- getSRA(search_terms = "breast cancer",
out_types = c("run", "study"), sra_con = sra_con)
全文検索:"submission" が "GEO" のエントリの検索rs <- getSRA(search_terms = "submission_center: GEO",
out_types = c("submission"), sra_con = sra_con)
全文検索:"study" に Carcinoから始まるワードが含まれるエントリの検索rs <- getSRA(search_terms = "Carcino*",
out_types = c("study"), sra_con = sra_con)
fastq:SRR000648とsRR000657のFastqファイルの取得getFastq(in_acc = c("SRR000648", "SRR000657"),
sra_con = sra_con, destdir = getwd())
IGVでのシーケンスの可視化:hg18 の chr1:1-1000 として表示exampleBams = file.path(system.file("extdata", package = "SRAdb"),
dir(system.file("extdata", package = "SRAdb"),
pattern = "bam$"))
sock <- IGVsocket()
IGVgenome(sock, "hg18")
IGVload(sock, exampleBams)
IGVgoto(sock, "chr1:1-1000")
IGVsnapshot(sock)
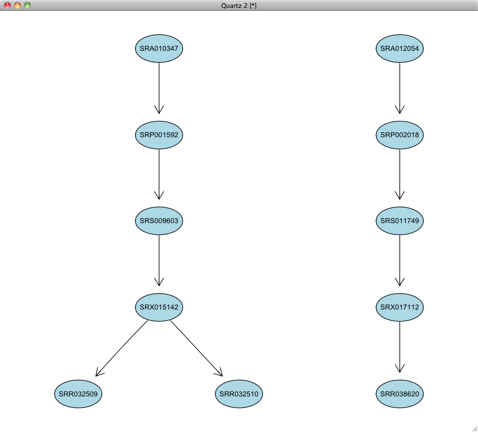
SRAエントリの可視化:"colon canver"を含むエントリacc <- getSRA(search_terms = "colon cancer",
out_types = c("sra"), sra_con = sra_con, acc_only = TRUE)
g <- entityGraph(acc)
attrs <- getDefaultAttrs(list(node = list(fillcolor = "lightblue",
shape = "ellipse")))
plot(g, attrs = attrs)
|
|
NCBI, EBI, DDBJ でのキーワード検索のリンクを調べてみました。"ChIP-Seq" で検索してみます。 NCBI SRA EBI SRA DDBJ DRA
|
|
DDBJ DRA でもキーワード検索が行えるようになりましたので、お知らせします。 |

