|
いつも参考にさせていただいています。 イントラで galaxy と UCSC Genome Browserを構築しているのですが、 galaxyで作成した複数のBAMデータをUCSC Genome Browser で並べて表示したい(Custom Trackに複数セットしたい) のですが、そのような連携のさせかたをご存知の方はいらっしゃいますでしょうか? 現在並べて表示する方法としては2パターンわかっているのですが、 もっと簡単に連携できないものかと思案しています。 パターン1:(bed,bedgraph,wig) パターン2:(BAM,bed,bedgraph,wig) よろしくお願いします。 ※hgt.customText の取得方法
|
|
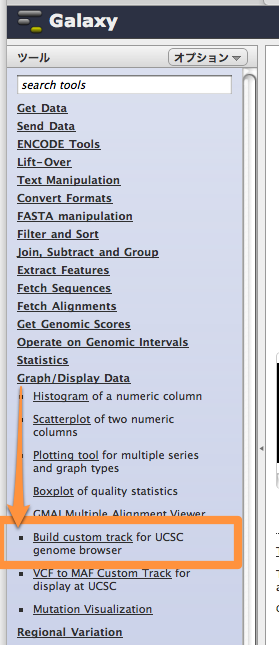
三つのヒストリアイテムをUCSCゲノムブラウザのカスタムトラックに表示する操作が Galaxy | Published Page | Screencasts の Finding promoters containing TAF1 binding sites identified from a CHiP-seq experiment にあります。また、そのシナリオは日本語で箇条書きした事があるのでご参考までに。 操作は、ツール:Graph/Display Data/Build custom track for UCSC genome browser をつかって、複数のヒストリアイテムからカスタムトラックを作成しています。
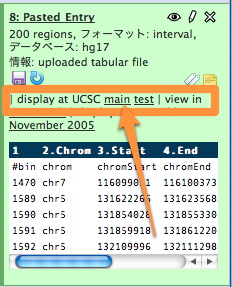
トラックの色やラベルを設定することができます。こうして作成したカスタムトラックをイントラのゲノムブラウザに送れば良いと思います。ヒストリアイテムの "display at UCSC main" のリンク先を genome.ucsc.edu からイントラ向きに変更すれば非常に簡単に連携することができるとおもいます。どこかの設定ファイルで変更できると思います。
回答ありがとうございます。 "Build Custom track" ツールでデータを指定してみたのですが、 BAMフォーマットのデータを指定することはできませんでした。 他に情報がありましたら引き続き、お願いします。 |